Verbundprojekt: TPMG Bielefeld |
| Technologieplattform Bioinformatik |
| Laufzeit: 01.04.10-31.12.13 |
| Verbundkoordinator: Dr. Alexander Goesmann, Universität Bielefeld, Bielefeld |
| Projektpartner: (alphabetische Reihenfolge) |
| Dr. Alexander Goesmann, Universität Bielefeld |
| Prof. Dr. Alfred Pühler, Universität Bielefeld |
| Die aktuelle Forschung auf dem Gebiet der Genom- und Metagenomanalysen wird derzeit insbesondere durch die ultraschnellen Hochdurchsatz-Sequenzierungstechnologien der Firmen 454 Life Sciences, Illumina Inc. und Applied Biosciences vorangetrieben. Bedeutendster Vorteil dieser Technologien ist neben einer dramatischen Reduzierung der Sequenzierkosten eine ebenso umfassende Senkung des erforderlichen experimentellen Aufwands. In zahlreichen Sequenzierprojekten konnten bereits die Früchte dieser neuen Technologien geerntet werden â eine Entwicklung die jedoch auch in einem enormen Anstieg bioinformatisch auszuwertender Sequenzdaten resultierte. Anforderungen an die Bioinformatik umfassen nicht nur die Assemblierung und funktionale Annotation pro- und eukaryotischer Sequenzdaten sondern darüber hinaus die vergleichende Analyse nahe-verwandter Arten und Stämme wie auch Studien zur Zusammensetzung mikrobieller Gemeinschaften verschiedener Ãkosysteme. |
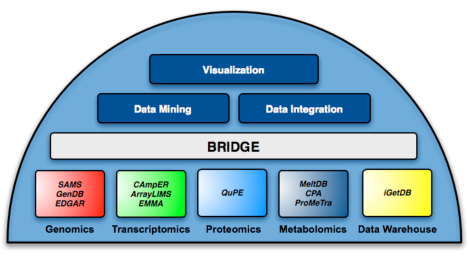 |
| Die Technologieplattform-Bioinformatik in Bielefeld blickt auf eine langjährige Expertise in der bioinformatischen Verarbeitung genomischer und postgenomischer Datensätze zurück. Bereits während der Förderinitiativen "GenoMik" und "GenoMik-Plus" des Bundesministeriums für Bildung und Forschung (BMBF) fungierte die Technologieplattform als zentral Bioinformatik-Ressource für Projektpartner. Im Rahmen der "GenoMik-Transfer-Initiative" wird jedem Kooperationspartner eine umfassende Infrastruktur zur Speicherung von Rohdaten und Analyseergebnissen zur Verfügung gestellt. Ein Hochverfügbarkeits-Rechencluster garantiert die zeitnahe und detaillierte Auswertung von Experimentaldaten. Hierzu bietet die Technologieplattform Zugriff auf ein umfassendes Portfolio bioinformatischer Werkzeuge und Softwareapplikation, die von der funktionalen Genomanalyse über die Metagenomik bis hin zu Transkriptom- Proteom und Metabolomanalysen reichen. Besonderes Augenmerk wird dabei stets auf die Gestaltung benutzerfreundlicher Oberflächen und eine leichte und verständliche Bedienbarkeit gelegt. Die Bielefelder Software-Palette umfasst derzeit i) GenDB zur Annotation und Pflege kompletter Genome (Meyer et al., 2003) , ii) SAMS als Sequenzanalyse und Verwaltungssystem (Bekel et al., 2009), iii) EDGAR für die vergleichende Analyse nahe-verwandter Genome (Blom et al., 2009), iv) EMMA zur Auswertung von Transkriptomdaten (Dondrup et al., 2009) sowie eine Fülle weiterer Applikationen aus den Bereichen Metagenomik, Proteomik und Metabolomik (http://www.cebitec.uni-bielefeld.de/cebitec/computational-genomics/home.html). Für Ihre Anfragen stehen wir gerne zur Verfügung â bitte wenden Sie sich an: Dr. Alexander Goesmann, eMail: agoesman (at) cebitec.uni-bielefeld.de. |
 |
Referenzen: |
| T. Bekel, K. Henckel, H. Küster, F. Meyer, V. Mittard Runte, H. Neuweger, D. Paarmann, O. Rupp, M. Zakrzewski, A. Pühler, J. Stoye, A. Goesmann (2009) The Sequence Analysis and Management System - SAMS-2.0: Data management and sequence analysis adapted to changing requirements from traditional sanger sequencing to ultrafast sequencing technologies. J Biotechnol 140(1-2): 3-12. |
| J. Blom, S. P. Albaum, D. Doppmeier, A. Pühler, F. J. Vorhölter, M. Zakrzewski, A. Goesmann (2009) EDGAR: A software framework for the comparative analysis of prokaryotic genomes. BMC Bioinformatics 10(1): 154. |
| M. Dondrup, S. P. Albaum, T. Griebel, K. Henckel, S. Jünemann, T. Kahlke, C. K. Kleindt, H. Küster, B. Linke, D. Mertens, V. Mittard Runte, H. Neuweger, K. J. Runte, A. Tauch, F. Tille, A. Pühler, A. Goesmann (2009) EMMA 2 - A MAGE-compliant system for the collaborative analysis and integration of microarray data. BMC Bioinformatics 10(1): 50. |
| F. Meyer, A. Goesmann, A.C. McHardy, D. Bartels, T. Bekel, J. Clausen, J. Kalinowski, B. Linke, O. Rupp, R. Giegerich, A. Pühler (2003) GenDB--an open source genome annotation system for prokaryote genomes. Nucleic Acids Res 31(8): 2187-95. |
GenoMik-Transfer - Anwendungsorientierte Genomforschung an Mikroorganismen
Neue Broschüre: Facetten der Genomforschung
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)



