Verbundprojekt: ExpresSys |
| Neue Expressionssysteme für industriell relevante Gene |
| Laufzeit: 01.02.10-30.11.13 |
| Verbundkoordinator: Prof. Dr. Wolfgang Liebl, Technische Universität München, Freising-Weihenstephan |
| Projektpartner: (alphabetische Reihenfolge) |
| Dr. Sonja-Verena Albers, Max-Planck-Institut für terrestrische Mikrobiologie |
| Prof. Dr. Garabed Antranikian, Technische Universität Hamburg-Harburg |
| Prof. Dr. Karl-Erich Jaeger, Heinrich-Heine-Universität Düsseldorf |
| Prof. Dr. Wolfgang Liebl, Technische Universität München |
| Prof. Dr. Rolf Müller, Helmholtz-Inst. f. Pharmazeutische Forschung Saarland |
| Prof. Dr. Jörg Pietruszka, Heinrich-Heine-Universität Düsseldorf |
| Prof. Dr. Bettina Siebers, Universität Duisburg-Essen |
| Prof. Dr. Wolfgang Streit, Universität Hamburg |
| Industriepartner: |
| BRAIN AG, Zwingenberg |
| Evonik Industries, Marl |
| Henkel KGaA, Düsseldorf |
| Süd-Chemie AG, München |
| Wacker Chemie AG, München |
 Dieser Projektverbund befasst sich mit Unzulänglichkeiten der üblicherweise eingesetzten Wirts-/Expressions-Systeme: Gene für wissenschaftlich oder industriell interessante Enzyme werden in Standardwirten wie E. coli oft nicht gut exprimiert. Weiterhin ist in funktionellen metagenomischen Screenings nur ein Bruchteil der immensen genetischen Diversität der Mikroorganismen in der Natur zugänglich, was vor allem an der begrenzten Fähigkeit der traditionellen Wirte zur Expression von Fremdgenen und der Schwierigkeit der Herstellung umfangreicher (meta)genomischer Genbanken in anderen Wirten als E. coli liegt. Dieser Projektverbund befasst sich mit Unzulänglichkeiten der üblicherweise eingesetzten Wirts-/Expressions-Systeme: Gene für wissenschaftlich oder industriell interessante Enzyme werden in Standardwirten wie E. coli oft nicht gut exprimiert. Weiterhin ist in funktionellen metagenomischen Screenings nur ein Bruchteil der immensen genetischen Diversität der Mikroorganismen in der Natur zugänglich, was vor allem an der begrenzten Fähigkeit der traditionellen Wirte zur Expression von Fremdgenen und der Schwierigkeit der Herstellung umfangreicher (meta)genomischer Genbanken in anderen Wirten als E. coli liegt. |
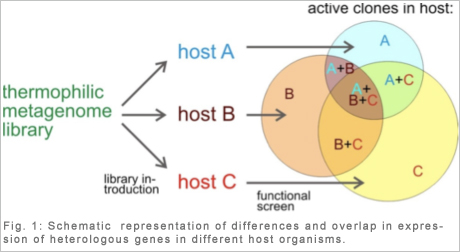 |
Deshalb werden neue Klonierungs- und Expressionswirte gebraucht für (i) die Steigerung der Detektionshäufigkeit der interessierenden Gene in funktionellen Hochdurchsatz-Screening-Strategien und (ii) die Ãberexpression von rekombinanten Proteinen. Wir haben vor, eine Expressionsplattform aus phylogenetisch verschiedenen Wirtsorganismen (Thermus thermophilus, Micrococcus luteus, Rhodobacter capsulatus, Myxococcus xanthus, Sulfolobus solfataricus und S. acidocaldarius) zu etablieren. Die wesentlichen Ziele des Forschungsverbunds sind:
|
 |
GenoMik-Transfer - Anwendungsorientierte Genomforschung an Mikroorganismen
Neue Broschüre: Facetten der Genomforschung
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)



