Verbundprojekt: FlexFit / Beschreibung Teilprojekt |
| Teilprojekt 5: Proteinschäden und ihre physiologische Rolle in Corynebacterium glutamicum bei verschiedenen Sauerstoffbedingungen |
| Laufzeit: 01.09.09-31.12.12 |
| Projektleiter: |
| Dr. Ansgar Poetsch, Ruhr-Universität Bochum |
| Dr. Christian Troetschel, Ruhr-Universität Bochum |
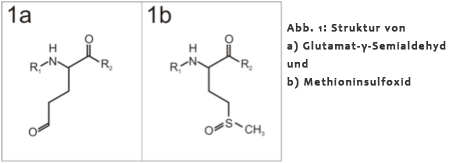
| Seitdem Sauerstoff in der Atmosphäre akkumulierte und auch in zellulären Prozessen wie der oxidativen Phosphorylierung verwendet wird, sind Organismen oxidativem Stress ausgesetzt. Ständig entstehen reaktive Sauerstoffspezies (ROS, reactive oxygen species), die alle wichtigen Biomoleküle in der Zelle schädigen können â DNA, Lipide und vor allem auch Proteine, die essentielle Schaltzentralen im Metabolismus darstellen. Durch die Bildung von ROS, wie z.B. dem Superoxid-Radikal, wird eine Vielzahl von Aminosäuren in den Proteinen modifiziert. Die meisten Modifikationen, wie etwa die Carbonylierung, bei der z.B. Glutamat-γ-Semialdehyd (s. Abb. 1a) aus Prolin und Arginin gebildet wird, sind irreversibel â eine Beseitigung des Schadens kann nur über den Abbau des betroffenen Enzyms erfolgen. Lediglich die beiden Schwefel-haltigen Aminosäuren Cystein und Methionin werden reversibel modifiziert - so kann das Methioninsulfoxid (s. Abb. 2) zum Beispiel durch entsprechende Reparaturenzyme wieder reduziert werden. |
 |
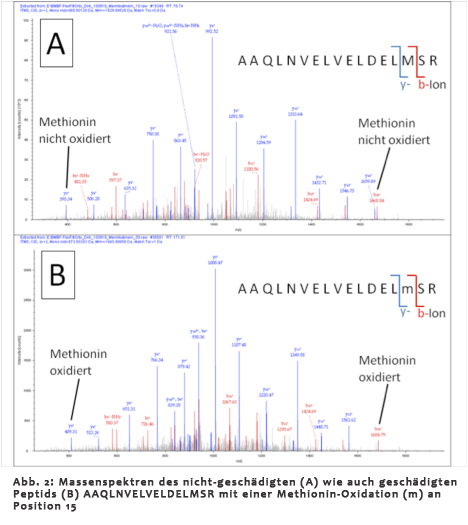
| Das Ziel dieses Teilprojekts ist zum einen, die Art und das Ausmaà der oxidativen Proteinmodifikationen in Corynebacterium glutamicum zu charakterisieren, die unter den gewählten, biotechnologisch relevanten Wachstumsbedingungen von Bedeutung sind. Dabei werden sowohl SDS-Gel basierte, wie z.B. der OxyblotTM zur Detektion von carbonylierten Proteinen, als auch Gel-freie Shotgun-Proteomik-Methoden eingesetzt. Mittels Massenspektrometrie kann anschlieÃend die (Teil-) Sequenz des Peptids (MS2-Spektrum, s. Abb. 2A und B) und damit die geschädigte Aminosäure identifiziert werden. Zum anderen wird weitergehend mittels Massenspektrometrie der Anteil der Modifizierung in Abhängigkeit von der Wachstumsbedingung quantifiziert. Auf Grundlage dieser Informationen soll die Aminosäuresequenz des geschädigten, nicht-funktionalen Proteins insoweit geändert werden, dass das Enzym in einem Produktionsstamm resistenter gegenüber oxidativem Stress ist. |
 |
GenoMik-Transfer - Anwendungsorientierte Genomforschung an Mikroorganismen
Neue Broschüre: Facetten der Genomforschung
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)



